| |
| ホーム | 概要 | 組織 | 研究 | 教育 | 活動 | アーカイブ | |
 |
環境ゲノミクス |
[ English | Japanese ] |
 |



 |
  |
 |
京都大学 化学研究所
バイオインフォマティクスセンター
教授
馬見塚 拓 |
生命科学研究は実験技術の進歩とあいまって個々の生体分子の解析から細胞内の生体分子全体の網羅的かつ統括的理解へと移りつつある。我々の研究は代謝パスウェイ等に代表される生体分子のネットワークに内在する生体内の分子メカニズムの解明である。具体的には、計算機科学や統計科学をベースとし、細胞あるいは組織レベルでの生体分子ネットワークの原理を体系的に解明するための計算論的技術の構築を行っている。特に、創薬科学を念頭においた場合には低分子化合物を含む生体分子ネットワークの解明が検討対象となる。我々が進めてきた関連研究の例を以下2点挙げる。
グラフ理論による化合物記述子の開発
グラフ理論において「木幅(tree-width)」と呼ばれるグラフの複雑さの尺度が知られている。木幅はグラフの木への近さを表す尺度であり、ノード数nのグラフに対し、木であれば1、環状の部分構造を持てば2、完全グラフであればn-1となる。そこで、化合物の化学式の平面構造をグラフとみなし、生体内化合物及びそれらの化学反応と木幅の関係から木幅による化合物記述の有用性を示した。より具体的には、木幅を変化させる反応の酵素種類は限られたEC番号を持つ酵素に集中する、また、代謝パスウェイをグラフとみなした場合に木幅を変化させる反応をこのグラフから除去するとグラフ結合性が大きく変化する、等が挙げられる。
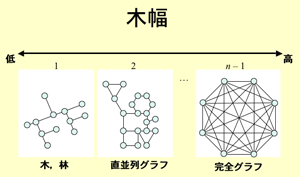 図1 木幅
図1 木幅
マイクロアレイデータからの代謝パスウェイの推定
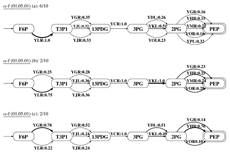
代謝パスウェイデータベースは文献の生体内化学反応情報から作られており、ある条件において各パスが有効か否かという情報は提供されないことが多い。そこで、既存のパスウェイに対してマイクロアレイデータにより生体内で実際に有効なパスを抽出する手法を構築した。提案手法は、パスウェイを混合マルコフモデルとみなしマイクロアレイデータからその確率パラメータを推定することにより有効パスを抽出する。モデルパスウェイとして解糖系の一部を用いた実験により有効パスの抽出のみならずパス上の遠距離相互作用(遠距離に位置する反応間における酵素の選択性)が抽出でき手法の有用性を確認した。
 |
|
 |
| 図2 得られた3種類の代謝パスパターン |
|
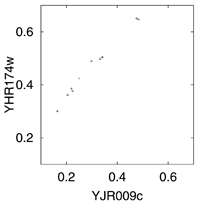
図3 T3P1→13PDGと2PG→PEP間の遠距離相互作用 |
|
|平成19年度|平成18年度|平成17年度|平成16年度|平成15年度|
※平成17年度より「環境ゲノミクス」へ移動 |
